化学実験、バイオ実験のノウハウなど、毎日の実験・分析に役立つ情報をお届け。

細胞の生命活動に不可欠なあらゆる機能は、ゲノムDNAから随時転写/合成されているRNA(トータルRNA:mRNA, tRNA, rRNA, 非コードRNAなど)によって厳密に制御されています。「どのようなRNA」が「どの程度の分子数」で存在しているかを知ることは、その細胞のリアルタイムの生命活動を明らかにする手がかりとなります。
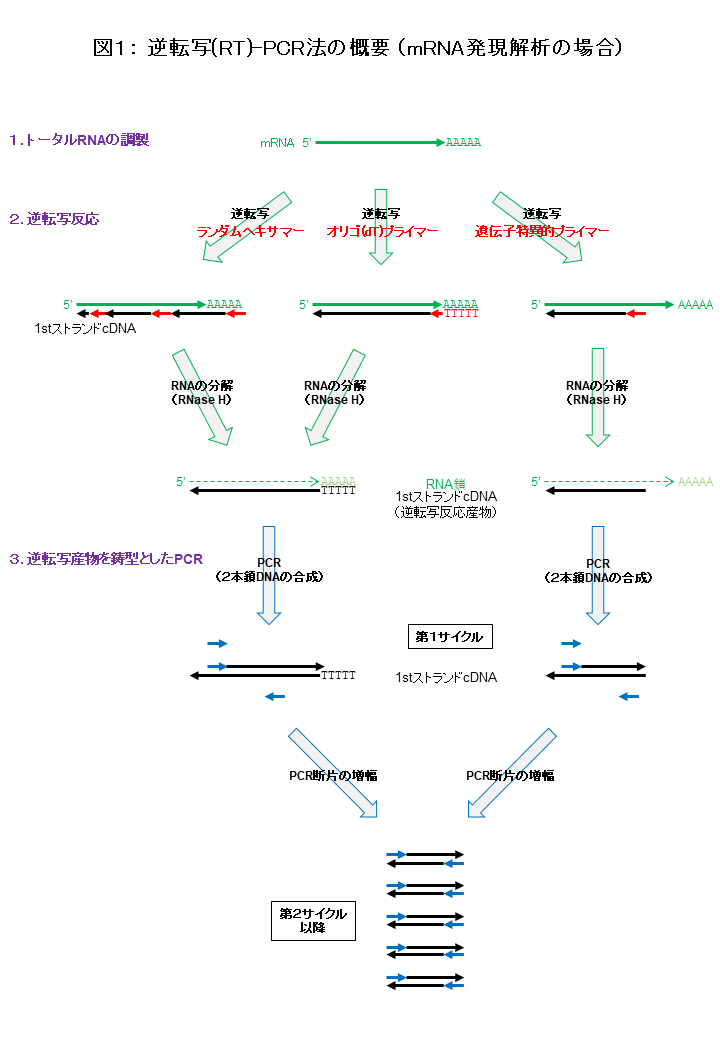
一方で、RNAは、細胞内や試験管内で極めて不安定な生化学的性質をもち、その検出・解析には、いくつかの技術的なポイントを押さえる必要があります。ここでは、逆転写反応(Reverse transcription (RT):RNAを鋳型としたDNA鎖の合成反応)を利用して、RNAの配列特異的な検出・解析を実現する「逆転写(RT)-PCR法」をご紹介します。RT-PCR法は、「1. トータルRNAの調製→ 2. 逆転写反応→ 3. 逆転写産物を鋳型としたPCR」の3ステップから構成されます[図]。
本項では、「培養細胞内RNAのRT-PCR解析」を例に、ステップごとに手順とポイントを説明していきます。
1. トータルRNAの調製
TRIzol試薬(ambion社)を用いた手順を例に、トータルRNAの調製法をご紹介します。TRIzolによる酸性抽出条件では、細胞由来のRNA/DNA/タンパク質はそれぞれ水相/フェノール相/中間相へと分配され、RNAをDNAやタンパク質から分離して得ることができます。
<実験のポイント>
●RNase(RNA分解酵素)の混入を防ぐ。
RNaseは非常に安定な酵素活性をもち、実験者の手、空気中の埃に付着する微生物、またはこれらが触れた実験器具などから混入します。
→RNAは、クリーンベンチ内で、使い捨ての手袋、滅菌した使い捨てのプラスチック器具・ピペット、RNaseフリーの器具を使用して扱う。
※TRIzol試薬は強力なタンパク質変性作用をもち、RNaseを失活できる。
●ゲノムDNAの反応系への混入を防ぐ。
ゲノムDNAは、逆転写反応に関係なくPCRの鋳型となり増幅産物を生成します。
→逆転写以降のステップで、混入ゲノムDNAを鋳型としたPCR増幅が生じてしまうと、cDNA由来のPCR産物との判別が困難になる(PCR結果の誤解を生む)。
<実験の手順>
※手袋・実験衣・眼の保護具を用いて、TRIzolによる皮膚や衣類の傷害・汚染を避ける。
細胞の溶解 → RNAの抽出
1. 培養細胞に直接TRIzolを加え、ピペッティングで溶解する。
●接着細胞の場合:培養ディッシュにTRIzolを加えて、溶解液をチューブに回収する。(培養面積10 cm2あたり1 mL程度)
●浮遊細胞の場合:遠心回収した細胞ペレットにTRIzolを加える。
(5~10×106/個の細胞に対して1 mL程度)
*TRIzol添加前のPBS等による細胞洗浄を控える(RNAの分解を避けるため)。
*細胞溶解に用いたTRIzolの体積を基準に、後に添加するクロロホルムやアルコールの体積が決まる。TRIzolの量が少ないと、分離抽出でDNAが混入するリスクが生じる。
2. 5分間のインキュベートを行う。(核酸-タンパク質複合体を変性する)
*粘性が高い場合は、26ゲージ針を数回通す。(ゲノムDNAを切断する)
*この溶解液の状態で、-60~-80℃で1ヶ月間の保存が可能。
3. クロロホルム(TRIzolの0.2倍量)を加える。
4. チューブを15秒間激しく振って混和し、2~3分間インキュベートする。
5. 2~8℃、15分間、12,000xg で遠心分離する。暗赤色のフェノール-クロロホルム相(下層)/中間相/無色の水相(上層)に分離されたことを確認する。
*水相はTRIzol添加体積の約60%になる。
6. 水相を新しいチューブに移す。
*発現量が僅かなRNA検体の場合や、PCR反応で用いるプライマー対が同一エキソンにアニールする場合には、DNase処理(RNaseフリー条件)を行い、混入ゲノムDNAを分解しておく。反応後のDNaseは、フェノール-クロロホルム抽出とエタノール沈殿によって除去する。
RNAの精製
7. イソプロピルアルコール(TRIzolの0.5倍量)を水相に加えて混和する。
*RNA沈殿が生成する。
*RNAの収量/沈殿量が少ない場合:水相に共沈殿キャリアとしてグリコーゲンを加えても良い。(5〜10 μg 程度, 4 mg/mL 未満, RNaseフリー条件)
8. 室温で10分間インキュベートする。
9. 2〜8℃、10分間、12,000xg で遠心分離し、上清を除去する。
*チューブ壁・底部にRNAのゲル状ペレットが得られる。
10. RNAペレットに75%エタノール(TRIzolと等量)を加える。
*-60〜-80℃で1年間の保存が可能。
11. 2〜8℃、5分間、7500xg で遠心分離し、上清を除去する。
12. RNAペレットを風乾により乾燥させる(5-15分間程度)。
*RNAぺレットは、完全に乾燥させると溶解度が著しく低下し、再溶解が困難になる。
13. RNAペレットにRNaseフリー水を加えて、ピペッティングで再溶解する。
14. 55〜60℃で10分間インキュベートする。
15. 吸光光度計でRNAの定量・検定を行う。
*1×106 個の培養細胞から得られるRNAの推定収量
●上皮細胞: 8〜15 μg
●線維芽細胞: 5〜7 μg
*A260/280比< 1.6の場合:RNAが完全に再溶解していない、細胞溶解に用いたTRIzolが少ない、細胞溶解後のインキュベートが不十分、水相へのフェノール相の混入などの可能性が考えられる。
*-60〜-80℃で数ヶ月間の保存が可能。
次回は、トータルRNAを鋳型とした「逆転写反応」を紹介します!
*監修
パーソルテンプスタッフ株式会社
研究開発事業本部(Chall-edge/チャレッジ)
研修講師(理学博士)
この記事は、理系研究職の方のキャリア支援を行うパーソルテンプスタッフ研究開発事業本部(Chall-edge/チャレッジ)がお届けする、実験ノウハウシリーズです。
過去の記事一覧:実験レシピシリーズ
関連記事Recommend
-

フローサイトメトリー(5/5) フローサイトメトリーを用いたセルソーティング
リケラボ実験レシピシリーズ
-

フローサイトメトリー(4/5) フローサイトメトリーを用いた細胞周期の解析
リケラボ実験レシピシリーズ
-

フローサイトメトリー(3/5) 細胞試料中に存在する生細胞と死細胞を計数するために
リケラボ実験レシピシリーズ
-

フローサイトメトリー(2/5) プロトコル編
リケラボ実験レシピシリーズ
-

フローサイトメトリー(1/5)概要と原理編
リケラボ実験レシピシリーズ
-

細胞の免疫蛍光染色 実践編(2/2)
リケラボ実験レシピシリーズ
-

細胞の免疫蛍光染色 基本編(1/2)
リケラボ実験レシピシリーズ
-

細胞内でRNA干渉を人為的に誘導するために RNA干渉による遺伝子発現の調節(2/2)
リケラボ実験レシピシリーズ
-

RNA干渉(RNA interfering, RNAi)とは? RNA干渉による遺伝子発現の調節(1/2)
リケラボ実験レシピシリーズ
-

免疫沈降法(Immunoprecipitation)による目的タンパク質の濃縮 基本から応用まで
リケラボ実験レシピシリーズ